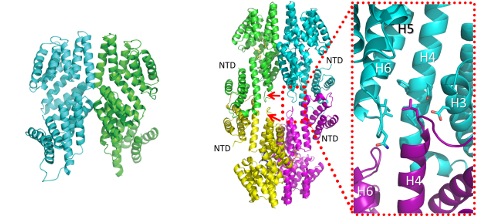

Izquierda: representación de la vista lateral de la estructura dimérica de RappLS20. Derecha: tetramerización RappLS20, vista lateral del tetrámero unido al péptido. Las flechas rojas indiquen los bucles que conectan las hélices H4 y H5. (C) Zoom del área alrededor del extremo N-terminal de la hélice H4, mostrando la inserción de esta hélice al monómero opuesto. La homotetramerización causada por las interacciones foot-2-foot de los NTD de RappLS20 proporciona una explicación para la activación de RcopLS20. En ausencia del péptido, los NTD se posicionan de manera que permiten la interacción con RcopLS20. No obstante, en unirse el péptido de señalización, los NTD se desplazan hacia el exterior, facilitando la formación del homotetrámero, provocando un cambio de la superficie de interacción de los NTD que ya no estaría disponible para las interacciones con RcopLS20. A la derecha, fotografía del equipo de ALBA.
Cerdanyola del Vallès, 20 de noviembre 2020 Las bacterias tienen mecanismos para establecer comunicación entre células. Esto es especialmente importante en la conjugación bacteriana, un proceso que les permite compartir material genético entre ellas. En muchos casos es usada por las bacterias para transferirse genes de resistencia a los antibióticos y otros factores de virulencia, aumentando de esta forma la difusión de la resistencia a antibióticos.
Ahora, un equipo de investigación de ALBA ha descubierto el mecanismo estructural mediante el cual dos proteínas, Rap y Rco, actúan juntas para regular la conjugación. Rco es una represora de la conjugación, mientras que Rap se une a Rco e impide entonces su represión de la conjugación, dando lugar a una activación del mecanismo de conjugación. Los principales resultados del estudio muestran que Rap contiene una zona donde un péptido corto se puede unir, produciéndole cambios estructurales que obligan a su tetramerización, liberando Rco para que bloquee la conjugación. La tetramerización se produce a través de una interacción que el equipo ha llamado "foot-2-foot", que difiere significativamente del modelo propuesto para otras proteínas de la familia Rap.
"Hemos demostrado por primera vez la interacción directa entre las proteínas Rap y Rco y hemos revelado el mecanismo por el cual la unión del péptido a Rap impide esta interacción Rap-Rco, reprimiendo el proceso de conjugación", explica Isidro Crespo, investigador en ALBA.
Nerea Bernardo, científica también de ALBA, añade que "mostramos como la proteína Rap sola tiene forma dimérica y que, cuando se une el péptido, forma tetrámeros dirigidos por el dominio N-terminal. Además, concluimos que Rap se une directamente a Rco en ausencia del péptido y, cuando este último es añadido, libera Rco de Rap. Esto permite a Rco unirse a la región promotora de genes cruciales para la conjugación, bloqueando su expresión."
La información de la base estructural del mecanismo de interacción se ha obtenido a través de la combinación de diversas técnicas de sincrotrón. La cristalografía de macromoléculas en la línea de luz XALOC de ALBA ha mostrado la estructura atómica de la proteína Rap y los cambios inducidos por la interacción peptídica, sugiriendo la tetramerización. Esto fue confirmado con estudios de SAXS (dispersión de rayos X a pequeños ángulos realizados en NCD-SWEET de ALBA y en BM29 del ESRF), SEC (cromatografía de exclusión por tamaño) y AUC (ultracentrifugación analítica). "Este enfoque del estudio combinando las cuatro técnicas ha sido crucial para descubrir el comportamiento de oligomerización de Rap y de los que interactúan con ella", comenta Roeland Boer, responsable de la línea de luz XALOC.
Finalmente, confirmaron que la tetramerización produce la disociación de Rco, liberándola debido a la interacción Rap-péptido. Los investigadores destacan que conocer el mecanismo de inhibición de la conjugación ayuda a encontrar estrategias clínicas para evitar la conjugación bacteriana y por tanto, la propagación de la resistencia a antibióticos. Los resultados muestran que la adición del péptido produce la disociación del complejo Rap-Rco, y Rco libre bloquea la conjugación. Por tanto, siguiendo esta estrategia, los medicamentos dirigidos a Rap podrían mejorar el bloqueo de la conjugación, evitando que las bacterias propaguen los genes de resistencia y otros factores de virulencia.
Los resultados representan otro avance más para la prevención de enfermedades, ya que estas proteínas se encuentran en muchos de los géneros bacterianos que son comensales o patógenos de los humanos, como Bacillus, Streptococcus y Enterococcus.
Referencia: Isidro Crespo, Nerea Bernardo, Andrés Miguel-Arribas, Praveen K Singh, Juan R Luque-Ortega, Carlos Alfonso, Marc Malfois, Wilfried J. J. Meijer, Dirk Roeland Boer. Inactivation of the dimeric RappLS20 anti-repressor of the conjugation operon is mediated by peptide-induced tetramerization. Nucleic Acids Research, 2020, 48, 14 8113–8127. DOI:10.1093/nar/gkaa540
El equipo de ALBA (Roeland, Nerea, Isidro) ha liderado el estudio, con ayuda del equipo de la línea NCD-SWEET (Marc Malfois) y colaboradores de Madrid, especialistas en conjugación bacteriana (Andrés Miguel-Arribas, Praveen K. Singh,Juan R. Luque-Ortega, Carlos Alfonso, Wilfried J.J. Meijer).




